ML 에서, 대부분 각 training data sample 각각이 수천~수백만개의 feature를 가지고 있다.
이렇게 feature가 많으면, 훈련이 느려질 뿐 아니라 좋은 성능을 내기 힘들다 (a.k.a 차원의 저주)
따라서 feature 수 즉 차원을 축소함으로서 성능 향상을 꾀할 수 있다. 또한 데이터 시각화에도 유용하다.
(ML에서의 경우 속도는 빨라지지만 일부 정보가 유실되므로 경우에 따라 성능이 나빠질 수 있다)
차원의 저주
고차원으로 갈수록 많은 공간을 가지고 있어 같은 단위 초입방체(hypercube)에 있더라도 평균거리가 크다.
즉, data가 멀리 떨어져있으므로 예측을 위해 더 많은 extrapolation(외삽)을 해야 하고 따라서 예측이 더 불안정하다.
-> 차원의 저주를 피하기 위해서는 training sample의 밀도가 높아질때까지 더 많은 sample을 모으는 것이지만,
실제로는 현실적으로 불가능할 정도로 엄청난 양의 sample을 모아야 하기에 한계 존재
-> 차원 축소
외삽
수학에서 원래의 관찰 범위를 넘어서서 다른 변수와의 관계에 기초하여 변수의 값을 추정하는 과정이다.
관찰된 값들 사이의 추정치를 만들어내는 보간법과 비슷하지만 보외법은 더 큰 불확실성과 무의미한 결과 생성에 대한 더 높은 위험에 종속된다. from 위키백과
투영(projection)
모든 traning sample이 고차원 space 내의 저차원 subspace에 놓여있으므로,
이 subspace에 수직으로 투영하여 타원 축소 가능

그러나 subspace가 뒤틀리거나 휘어있는 경우도 많기에, projection이 항상 좋지는 않음
e.g. 스위스롤 dataset
manifold
고차원 공간에서 휘어지거나 뒤틀린 2D 모양을 manifold라고 한다.

위와 같은 스위스롤 dataset는 manifold의 한 예시이다. 이 경우 2차원 초평면(d=2)으로 보일 수 있는 3차원 공간(n=3)의 데이터셋이다.
manifold learning
training sample에 놓여져있는 manifold를 모델링하는 것으로, 많은 차원 축소 알고리즘의 작동 원리이다.
이는 대부분 실제 고차원 dataset이 낮은 저차원 manifold에 가깝게 놓여져있다는 manifold hypothesis에 근거한다.
하지만 항상 저차원 dataset이 manifold에서 더 간단하게 표현되지는 않는다.
차원 축소 방법
PCA (principal component analysis, 주성분분석)
데이터에 가장 가까운 hyperplane을 정의한 후 데이터에 이 평면을 projection 한다.
이때, 올바른 hyperplane을 선택하는 것이 중요한데,
다른 방향으로 투영하는 것보다 분산이 최대로 보존되는 축 을 선택하면 정보가 가장 적게 손실된다.
(즉, 원본 dataset과 투영된 것 사이의 MSE를 최소화하는 축)
이렇게 선택한 것이 첫번째 PC(주성분)이 되고,
이후 첫번째 축에 직교하고 남은 분산들을 최대한 보존하는 축을 찾아 두번째 PC,
이전 두 축에 직교하는 세번째 축을 세번째 PC, … 이렇게 i번째 PC를 구할 수 있다.
이때 각 PC의 방향은 일정하지 않다.
또한 traning set를 조금 섞은 후 PCA를 적용하면, 새로운 PC 중 일부가 원래 PC와 반대방향일 수 있다
그러나 일반적으로는 같은 축에 놓여 같은 평면을 구성한다.
SVD (singular value dicomposition)
trainig set의 PCA는 SVD로 분해한다.

코드는 아래와 같다.
X_centered = X - X.mean(axis =0)
U, s, Vt = np.lianlg.svd(X_centered)
c1 = Vt.T[:,0]
c2 = Vt.T[:,1]
이렇게 PC를 다 추출했다면, 처음 d개의 PC로 정의한 hyperplane에 투영하여 데이터셋의 차원을 모두 d차원으로 축소 가능하다.
(이때, hyperplane은 분산을 최대로 보전하는 projection임을 보장한다.)
W2 = Vt.T[:,2] # 첫 2개의 PC로 정의된 평면
X2D = X_centered.dot(W2) # training set를 projection
이 모든 PCA는 아래와 같이 sklearn으로 처리 가능하다.
from sklearn.decomposition import PCA
pca = PCA(n_components = 2)
X2D = pca.fit_transform(X)
이때 pca.explained_variance_ratio_를 통해 PC의 explaned variance ratio를 알 수 있다.
이 비율은 각 PC의 축을 따라 있는 데이터셋의 분산 비율을 나타낸다.
예를 들어
>>> pca.explained_variance_ratio_
array([0.84248607, 0.14631839])
위에서 데이터셋 분산의 84.2%가 첫 번째 PC를 따라 놓여 있고 14.6%가 두 번째 PC를 따라 놓여있음을 의미한다.
이때 축소할 차원의 수(d)를 임의로 정하기보다,
아래 코드처럼 충분한 분산(e.g. 95% 이상)이 될때까지 더해야할 차원의 수를 선택하는 것이 좋다.
pca = PCA()
pca.fit(X_train)
cumsum = np.cumsum(pca.explained_variance_ratio_)
d = np.argmax(cumsum >= 0.95) + 1
또는 차원수에 따른 설명된 분산(0~1)을 그래프로 그려 확인하기도 한다.
데이터 시각화의 경우에는 2,3차원으로 줄이는 것이 일반적이다.
또한 아래와 같이, PCA projection을 반대로 적용하여 원래 차원으로 되돌리기도 한다.
pca = PCA(n_components = 154)
X_reduced = pca.fit_transform(X_train)
X_recovered = pca.inverse_transform(X_reduced)
randomized PCA
incremental PCA
훈련 세트를 미니배치로 나눈 뒤 IPCA 알고리즘에 한 번에 하나씩 주입하여 PCA를 적용할수도 있다.
from sklearn.decomposition import IncrementalPCA
n_batches = 100
inc_pca = IncrementalPCA(n_components=154)
for X_batch in np.array_split(X_train, n_batches):
inc_pca.partial_fit(X_batch)
X_reduced = inc_pca.transform(X_train)
Kernel PCA (kPCA)
kernal trick을 PCA에 적용하여 복잡한 비선형 projection을 수행할 수 있는데, 이를 kPCA라고 한다.
이는 projection된 후에 샘플의 군집을 유지하거나, 꼬인 manifold에 가까운 데이터셋을 펼칠 때도 유용하다.
from sklearn.decomposition import KernelPCA
rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.04)
X_reduced = rbf_pca.fit_transform(X)
kPCA는 unsupervised learning 이기 때문에 좋은 kernal, hyperparameter를 선택하기 위한 명확한 성능 측정 기준이 없다.
하지만 kernal trick은 supervised learning 의 전처리로 사용되는 경우가 많으므로, gridsearch를 통해 가장 성능 좋은 kernal, hyperparameter를 찾을 수 있다. 아래 코드를 참고하자.
from sklearn.model_selection import GridSearchCV
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
clf = Pipeline([
("kpca", KernelPCA(n_components=2)),
("log_reg", LogisticRegression())
])
param_grid = [{
"kpca__gamma": np.linspace(0.03, 0.05, 10),
"kpca__kernel": ["rbf", "sigmoid"]
}]
grid_search = GridSearchCV(clf, param_grid, cv=3)
grid_search.fit(X, y)
이러한 kernal trick 덕분에, kPCA의 결과는
feature map을 이용해 traning set를 무한 차원의 feature space에 mapping한 후 변환된 dataset을 PCA를 이용해 2D로 projection 한것과 수학적으로 동일하다.
재구성 원상 (pre-images)
축소된 공간에 있는 샘플에 대해 선형 PCA를 역전시키면 재구성된 데이터 포인트는 원본 공간이 아닌 feature space에 놓이게 되는데,
이는 무한 차원이기 때문에 재구성된 포인트를 계산할 수 없고 재구성에 따른 실제 에러를 계산할 수 없다.
다행히 재구성된 포인트에 가깝게 매핑된 원본 공간의 포인트를 찾을 수 있는데, 이를 재구성 원상이라고 한다.
rbf_pca = KernelPCA(n_components = 2, kernel="rbf", gamma=0.0433,
fit_inverse_transform=True)
X_reduced = rbf_pca.fit_transform(X)
X_preimage = rbf_pca.inverse_transform(X_reduced)
LLE (locally linear embedding)
비선형 차원 축소 기술로, projection에 의존하지 않는 manifold learning 이다.
먼저 각 훈련 샘플이 가장 가까운 이웃에 얼마나 선형적으로 연관되어 있는지 측정한 후,
(각 훈련 샘플 xi에 대해 가장 가까운 k개의 샘플을 찾은 후, 이 이웃에 대한 선형함수로 xi를 재구성한다.
즉, 위 식에서 w가 최소가 되도록 하는 것)
국부적인 관계가 가장 잘 보존되는 훈련 세트의 저차원 표현을 찾는다.
이 방법은 잡음이 너무 많지 않은 경우 꼬인 매니폴드를 펼치는 데 잘 작동하지만, 대량의 데이터셋에 적용하기는 힘들다.
from sklearn.manifold import LocallyLinearEmbedding
lle = LocallyLinearEmbedding(n_components=2, n_neighbors=10)
X_reduced = lle.fit_transform(X)
이 외에도 random projection, MDS(multidimensional scaling), Isomap, t-SNE, LDA등을 사용한다.
이 중 t-SNE, LDA에 대해 조금 더 살펴보자.
t-SNE (티스니)
비슷한 샘플은 가까이, 비슷하지 않은 샘플은 멀리 떨어지도록 하면서 차원을 축소한다.
주로 시각화에 많이 사용되며 특히 고차원 공간에 있는 샘플의 군집을 시각화할 때 사용된다.
import matplotlib.pyplot as plt
from sklearn.manifold import TSNE
model = TSNE(learning_rate=100)
transformed = model.fit_transform(feature)
xs = transformed[:,0]
ys = transformed[:,1]
plt.scatter(xs,ys,c=labels)
plt.show()
#출처: https://bcho.tistory.com/1210 [조대협의 블로그]
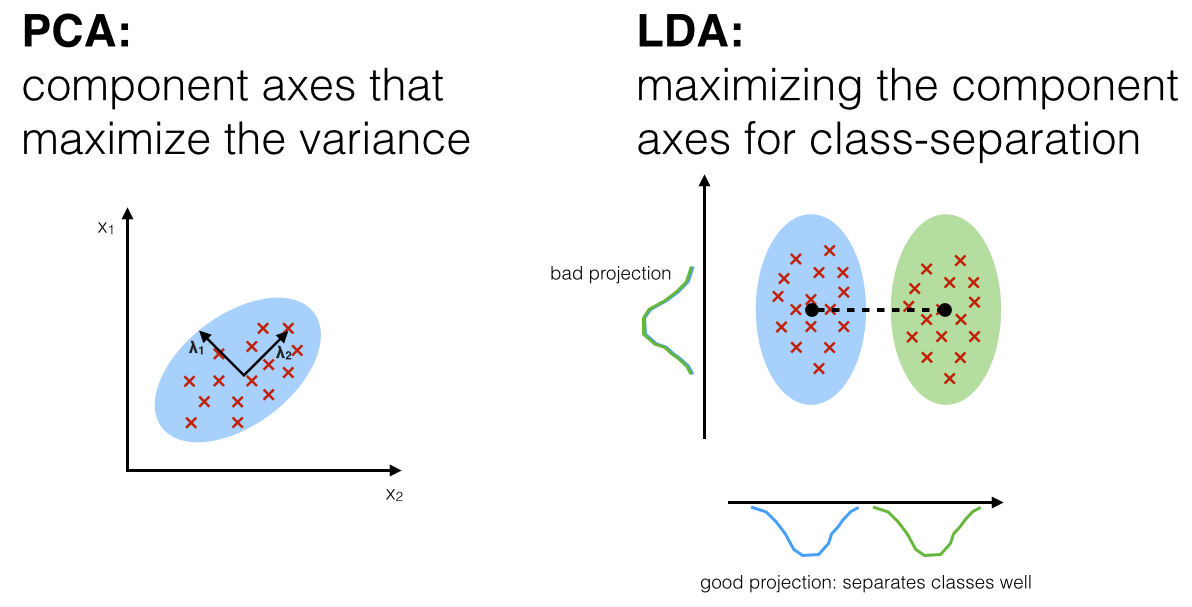
LDA (linear discriminant analysis)
훈련 과정에서 클래스 사이를 가장 잘 구분하는 축을 학습하여, 데이터가 투영되는 초평면을 정의하는 데 사용할 수 있다.
projection을 통해 가능한 한 클래스를 멀리 떨어지게 유지시키므로 분류 알고리즘을 적용하기 전에 차원을 축소시키는 데 좋다.
import numpy as np
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
clf = LinearDiscriminantAnalysis()
clf.fit(X, y)
LDA(Latent Dirichlet allocation)
이 LDA는 topic modeling 에서 쓰이는 알고리즘이다.
주어진 문서에 대하여 각 문서에 어떤 주제들이 존재하는지를 서술하는 대한 확률적 토픽 모델 기법 중 하나이다.
미리 알고 있는 주제별 단어수 분포를 바탕으로, 주어진 문서에서 발견된 단어수 분포를 분석함으로써 해당 문서가 어떤 주제들을 함께 다루고 있을지를 예측할 수 있다. from wikipedia

